이 콘텐츠는 사이트 회원 전용입니다. 기존의 사용자라면 로그인 하세요. 새 사용자는 아래에서 회원가입 할 수 있습니다.
3.5세대 유전자 가위(CRISPR/Cpf1) 논문 분석 2
- 펭 장 교수 팀, 새로운 CRISPR/Cpf1의 발견과 인간 세포대상 연구결과(2015)
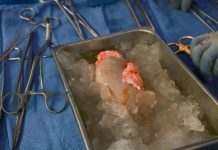
요약: 생명과학자들은 최근 3세대를 넘어 보다 정확하고 표적에 특이적인 3.5세대 유전자 가위(CRISPR/Cpf1)를 발견하고 개발했다. 인간세포와 동식물세포의 유전자를 마음대로 교정하는데(Editing) 사용한다. 표적 DNA를 자른 후 세포 내 복구 시스템에 의해 다시 연결되는 과정에서 유전자 교정과 원하는 변이가 일어난다. 이 방식을 활용해 암과 AIDS 등뿐만 아니라 더 나아가 희귀난치병 치료나 작물•가축개량•미래식량(Clean meat)






















![[보고서] 딥씨크 출현과 한국 AI 미래전략](http://itnews.or.kr/wp-content/uploads/2025/02/7-218x150.png)

![[2024 애플 동향] ③ 애플카 ‘프로젝트 타이탄’의 실패 원인](http://itnews.or.kr/wp-content/uploads/2024/03/1-2-218x150.png)
![[2024 애플 동향] ② 애플카 ‘프로젝트 타이탄’ 10년간 의 여정](http://itnews.or.kr/wp-content/uploads/2024/03/4-218x150.png)
![[2024 애플 동향] ① 애플 프로젝트 타이탄’ 종료](http://itnews.or.kr/wp-content/uploads/2024/03/Midjourney-218x150.png)






Comments are closed.